728x90

머신러닝 복습
비지도학습
- 레이블이나 명시적인 결과 없이 입력 데이터의 구조나 패턴을 발견하고 모델링하는 방법
비지도 학습만으로 끝나지 않고 후속 작업 필요
- 차원 축소: 고차원 데이터를 축소하여 새로운 feature 생성
- Clustering: 고객별 군집 생성
- 이상탐지: 정상 데이터 범위 지정
1. 차원 축소
- 고차원 데이터는 많은 특성(feature)을 가지고 있기 때문에 데이터를 시각화하거나 분석하기 어려움
- 또한, 차원이 증가함에 따라 데이터의 복잡성도 증가 및 학습 알고리즘의 성능이 저하됨(차원의 저주)
- 따라서 차원 축소는 데이터의 복잡성을 줄이고 효율적인 분석을 가능하게 함
- ex) 주성분 분석(PCA), t-SNE
주성분 분석(PCA)
- 데이터를 새로운 축으로 변환하여 가장 많은 분산을 보존하는 주성분을 찾아내는 방법
- 스케일링 필요
- PCA 절차
- 학습 데이터셋에서 분산이 최대인 첫번째 축(axis)을 찾음.
- 첫번째 축과 직교(orthogonal)하면서 분산이 최대인 두 번째 축을 찾음.
- 첫 번째 축과 두 번째 축에 직교하고 분산이 최대인 세 번째 축을 찾음.
- ①~③과 같은 방법으로 데이터셋의 차원 만큼의 축을 찾음.
# 주성분 분석 선언
# 주성분 1개짜리
pca1 = PCA(n_components=1)
x_pc1 = pca1.fit_transform(x_train)
# 주성분 2개짜리
pca2 = PCA(n_components=2)
x_pc2= pca2.fit_transform(x_train)
# 주성분 3개짜리
pca3 = PCA(n_components=3)
x_pc3= pca3.fit_transform(x_train)
# 각 주성분 결과 상위 3개 확인
print(x_pc1[:3])
print('-'*50)
print(x_pc2[:3])
print('-'*50)
print(x_pc3[:3])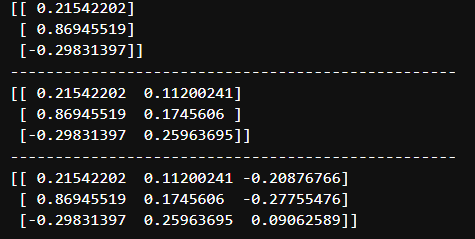
주성분의 개수 정하기
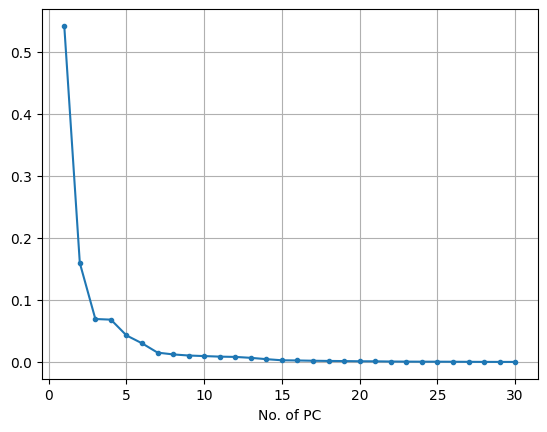
# 주성분 개수에 따른 주성분의 설명된 분산 비율 시각화
plt.plot(range(1,n+1), pca.explained_variance_ratio_, marker = '.')
plt.xlabel('No. of PC')
plt.grid()
plt.show()- .explained_variance_ratio_: 각 주성분이 설명하는 분산의 비율
Elbow Method
2. 클러스터링
- Cluster를 서로 유사한 그룹으로 분할하는 과정
- ex) K-Means, DBSCAN
K-Means
- K개의 평균으로 부터 거리를 계산하고, 가까운 평균으로 묶어 Cluster를 나누는 방식
- k-means 절차
- 클러스터의 개수 지정(k)
- 그룹의 중심 점(mean)이 무작위로 선택됨
- 임의로 선택된 중심 점과 각 점 간의 거리를 계산해서 가장 가까운 중심점의 그룹(군집)으로 선택됨
- 선택된 그룹의 점들을 기준으로 중심점을 계산해서 찾고,
- 중심점의 변화가 거의 없을 때까지 ⑤ ③~④를 반복
# k means 학습
model = KMeans(n_clusters= 2, n_init = 'auto')
model.fit(x)
# 예측
pred = model.predict(x)
print(pred)
적정 K 값 찾기
1. Inertia value
- 군집화가 된 후에, 각 중심점에서 군집의 데이터 간의 거리를 합산한 값
# inertia값 확인
model.inertia_
# k의 갯수에 따라 각 점과의 거리를 계산하여 적정한 k 찾기
kvalues = range(1, 10)
inertias = []
for k in kvalues:
model = KMeans(n_clusters=k, n_init = 'auto')
model.fit(x)
inertias.append(model.inertia_)# 시각화
plt.figure(figsize = (8, 6))
plt.plot(kvalues, inertias, marker='o')
plt.xlabel('number of clusters, k')
plt.ylabel('inertia')
plt.grid()
plt.show()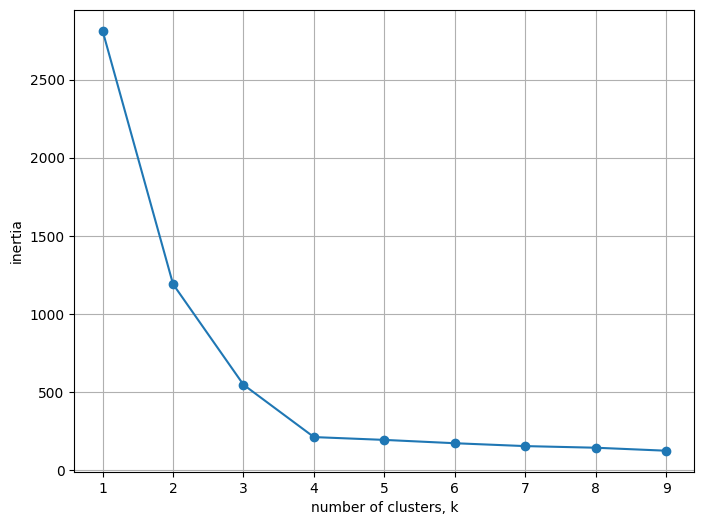
2. 실루엣 점수
| 실루엣 점수 | 결과 해석 |
| 1에 근접 | 클러스터 간 거리는 멀고, 클러스터 내부의 거리는 가까움(적절) |
| 0 에 근접 | 클러스터 간 거리와 클러스터 내부 거리가 비슷(클러스터가 중첩) |
| -1 에 근접 | 클러스터링 결과가 부적절 |
from sklearn.metrics import silhouette_score
# 클러스터 개수에 따른 실루엣 점수를 저장할 리스트
kvalues = range(2, 10) # 최소 2개 이상이어야 함.
sil_score = []
for k in kvalues:
# KMeans 모델 생성
model = KMeans(n_clusters=k, n_init = 'auto')
# 모델을 학습하고 예측
pred = model.fit_predict(x)
# 실루엣 점수 계산
sil_score.append(silhouette_score(x, pred))# 실루엣 점수 시각화
plt.figure(figsize = (8, 6))
plt.plot(kvalues, sil_score, marker='o')
plt.xlabel('n_clusters')
plt.ylabel('Silhouette Score')
plt.grid()
plt.show()
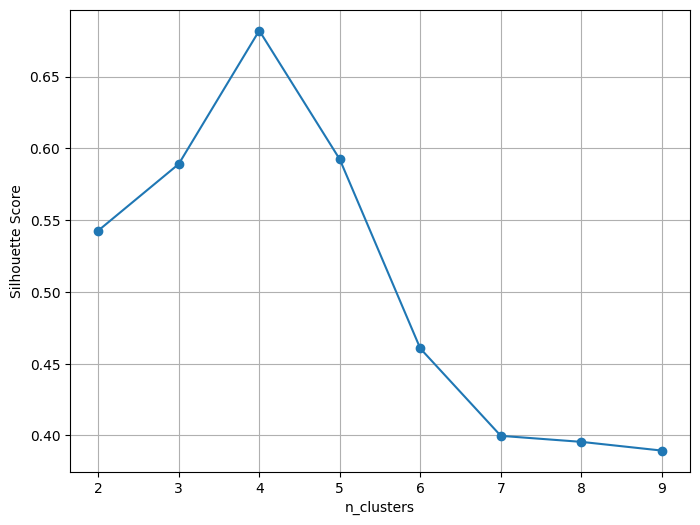
k 값에 따라 모델을 생성하고 그래프 그리기 함수
def k_means_plot(x, y, k) :
# 모델 생성
model = KMeans(n_clusters= k, n_init = 'auto')
model.fit(x)
pred = model.predict(x)
# 군집 결과와 원본 데이터 합치기(concat)
pred = pd.DataFrame(pred, columns = ['predicted'])
result = pd.concat([x, pred, y], axis = 1)
# 중앙(평균) 값 뽑기
centers = pd.DataFrame(model.cluster_centers_, columns=['x1','x2'])
# 그래프 그리기
plt.figure(figsize = (8,6))
plt.scatter(result['x1'],result['x2'],c=result['predicted'],alpha=0.5)
plt.scatter(centers['x1'], centers['x2'], s=50,marker='D',c='r')
plt.grid()
plt.show()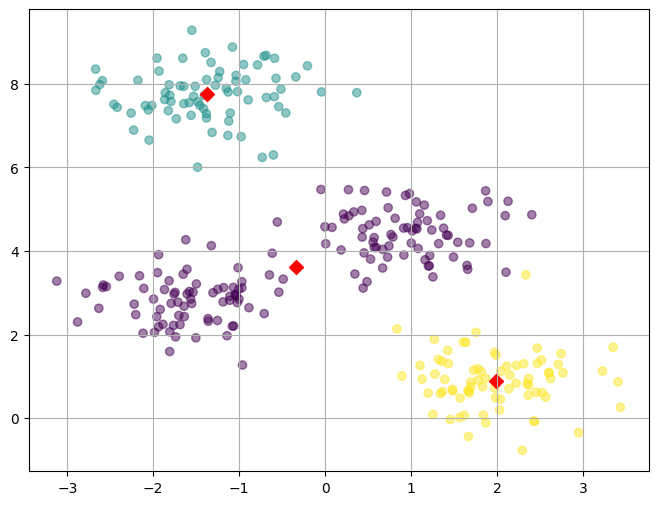
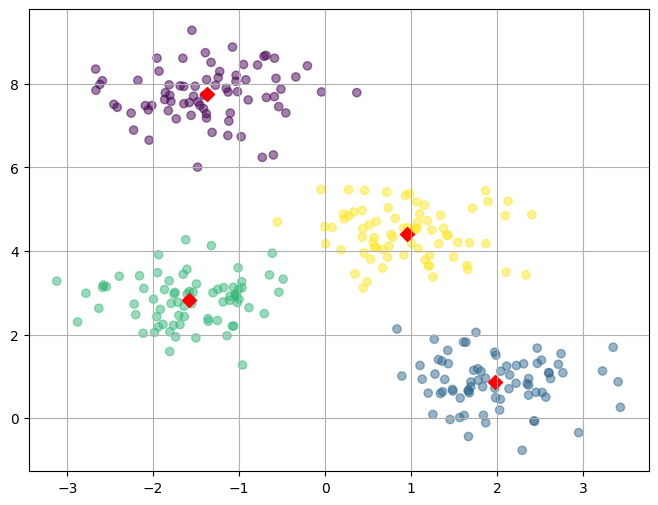
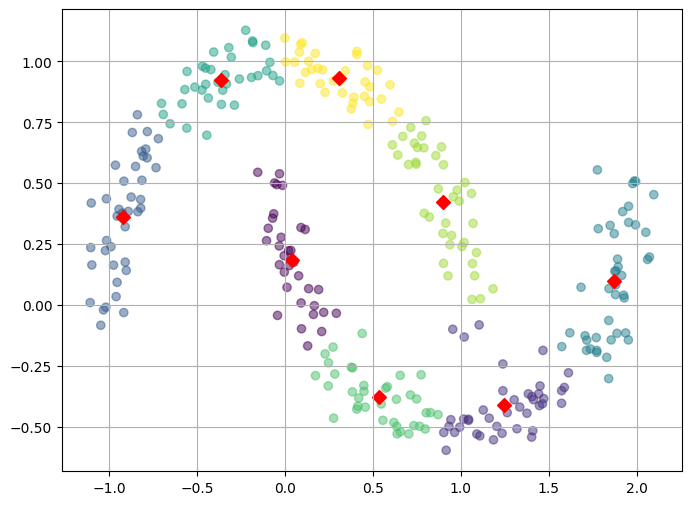
K-Means의 한계
- 중심점에서의 거리로 군집화
- 즉, 클러스터가 원형으로 분포되어 있는 경우에는 잘 작동하지만, 아래와 같이 비선형적인 형태의 클러스터를 잘 찾지 못할 수 있음
- DBSCAN으로 단점 보완이 가능하나 대체적으로 K-Means 사용

728x90

